DFG Heisenbergprogramm LE 3358/4-1 „DiaLReDox - Die Regulierung der photosynthetischen Lichtreaktion, lichtabhängiges retrogrades Redoxsignaling und Redoxdynamiken in der Diatomee Phaeodactylum tricornutum“
und DFG-Sachbeihilfeprogramm LE3358/5-1 “Identifizierung molekularer Signaltransduktionskomponenten des retrograden Redoxsignalings und Auflösung kompartimenteller Redoxdynamiken in der Kieselalge Phaeodactylum tricornutum”
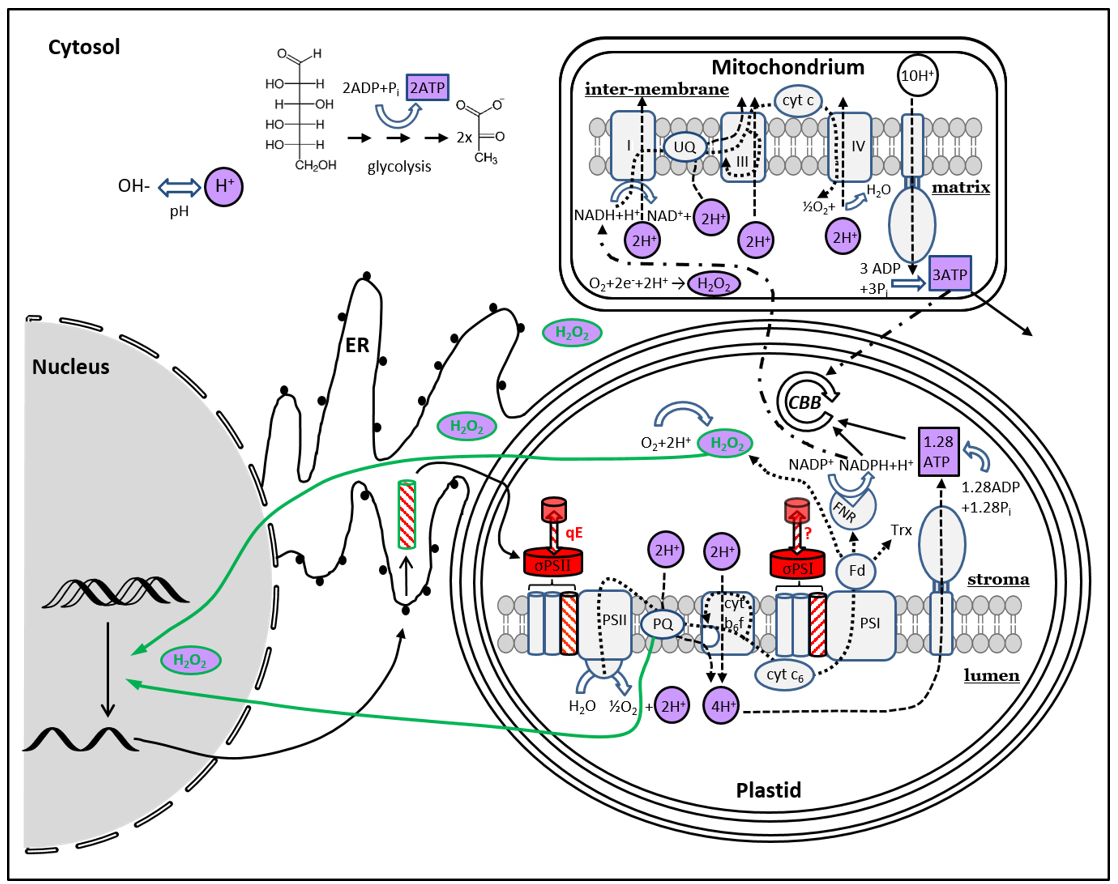
Diatomeen besitzen komplexe Plastiden, welche sie über sekundäre Endosymbiose aus einer Rotalge gewonnen haben. Daher besteht ihre Plastidenhülle aus vier Membranen, gegenüber deren zwei in Grünalgen und Gefäßpflanzen. Vor über zehn Jahren haben wir die Existenz von lichtabhängigem retrograden Redoxsignaling - die Steuerung der Kerngenexpression über einen Stimulus aus dem Plastiden - in der Diatomee Phaeodactylum tricornutum nachgewiesen. Dabei identifizierten wir den Redoxzustand des Plastochinonpools als Ausgangspunkt dieser Signalkaskade. Dieser ist nur einer von vielen sich dynamisch und lichtabhängig verändernden Redoxkomponenten in der Diatomeenzelle. Zum Beispiel sind pH, ATP und H2O2 von den Lichtbedingungen abhängig und werden potentiell vom retrograden Signaling beeinflusst bzw. steuern es selbst. Ihre kompartimentellen Dynamiken sind in Diatomeen völlig unverstanden.
In diesem Projekt wollen wir molekulare Komponenten des retrograden Redoxsignalings in Diatomeen enthüllen und Einsichten in die Signalkaskade, die im Plastiden entspringt und im Zellkern endet, gewinnen. Außerdem interessieren uns die lichtabhängigen kompartimentellen Redoxdynamiken des pHs, von ATP und von H2O2, besonders im Hinblick auf das Zusammenspiel der photosynthetischen Lichtreaktion mit der mitochondrialen Respiration.
Methodisch nutzen wir Transkriptomanalysen, Hefe-1-Hybrid-Assays, Fluoreszenzproteinbasierte Redoxsensoren, Fluoreszenzmikroskopie und -spektroskopie, und Genomediting.
Zusammen mit der dynamischen Regulation der Lichtreaktion, welche im DFG-Projekt LE3358/3-2 untersucht wird, werden die Ergebnisse dieses Projektes ein ganzheitlicheres Verständnis der Photophysiologie ermöglichen. Damit können neue Modelle bezüglich der Lichtakklimatisierung in Diatomeen, aber auch darüber hinaus in anderen Taxa etabliert werden.
DFG-Sachbeihilfeprogramm LE3358/3-2 „Die molekularen Grundlagen der schnellen Photoprotektion und ihr Einfluss auf die kompetitive Fitness in Phaeodactylum tricornutum“

Diatomeen sind in fast allen aquatischen Habitaten sehr erfolgreich. Als ein wesentlicher Grund dafür wird ihre ausgeprägte Photoprotektionskapazität basierend auf qE (energy dependent fluorescence quenching) angenommen. Während die Bedeutung des Carotinoids Diatoxanthin, welches im Xanthophyllzyklus während Starklichtexposition gebildet wird, für qE lange bekannt ist, wurde die Relevanz der Lhcx-Proteine für qE erst vor einigen Jahren enthüllt. Wir konnten hier zeigen, dass Lhcx1, x2 und x3, nicht aber Lhcx4, qE in Phaeodactylum tricornutum vermitteln.
In diesem Projekt wollen wir den qE-Mechanismus hinsichtlich seiner molekularen Komponenten genauer untersuchen. Dabei interessieren uns die Aminosäuren und Peptidmotive, die essentiell sind um ein Lhcx-Protein von einem klassischen Lichtsammelprotein funktionell zu unterscheiden. Außerdem wollen wir die Interaktionspartner der Lhcx-Proteine in der Thylakoidmembran aufspüren, denn bisher gibt es nur Indizien, wo dieser Mechanismus des qEs genau stattfindet. Und schließlich führen wir umfangreiche Wachstumsexperimente unter wechselnden Stressbedingungen mit verschiedenen Lhcx-Mutanten durch, um den tatsächlichen Einfluss des qEs unter naturnah simulierten Bedingungen zu enthüllen.
Methodisch verwenden wir reverse Genetik und Genomediting, verschiedene biochemische Ansätze sowie eine breite Palette photophysiologischer Geräte.
Unsere Ergebnisse integrieren sich in das generelle Ziel des Heisenbergprogramms, dem hochaufgelösten und detaillierten Verständnis der molekularen und physiologischen Antwort auf Lichtstress in Diatomeen.
